科学家用分子动力学和AlphaFold,揭示了转运蛋白的未解结构
- 创业科技
- 2024-02-21
- 3
- 更新:2024-02-21 11:59:14
编辑 | 萝卜皮
转运蛋白改变其构象以携带其底物穿过细胞膜。构象动力学对于理解运输功能至关重要。
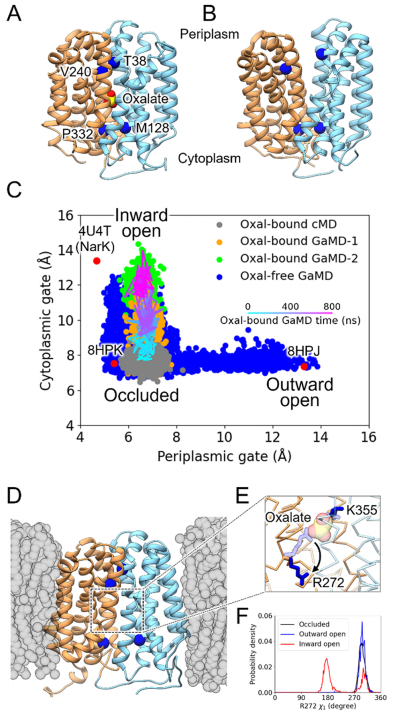
日本国立自然科学研究院(National Institutes of Natural Sciences)和冈山大学(Okayama University)的合作团队研究了草酸转运蛋白(OxlT),这是一种来自产酸草酸杆菌的草酸:甲酸逆向转运蛋白,对于避免肾结石形成具有重要意义。OxlT 的原子结构最近已在向外开放和封闭状态下得到解决。然而,向内开放的构象仍然缺失,阻碍了研究人员对转运蛋白的完整理解。
在最新的研究中,该团队进行了高斯加速分子动力学模拟,对 OxlT 的广泛构象空间进行采样,并成功预测了向内开放的构象,其中细胞质底物甲酸盐结合优先于草酸盐结合。
该团队还确定了向内开放构象的关键相互作用。AlphaFold2 结构预测对结果进行了补充。尽管 AlphaFold2 仅预测向外开放构象中的 OxlT,但所识别的关键残基的突变使其部分地预测向内开放构象,从而识别出可能的状态转移突变。
该研究以「Accelerated Molecular Dynamics and AlphaFold Uncover a Missing Conformational State of Transporter Protein OxlT」为题,于 2024 年 1 月 12 日发布在《The Journal of Physical Chemistry Letters》。
蛋白质是生命的基石,在每个生物体中发挥重要功能。转运蛋白(如 OxlT)尤其重要,因为它们携带重要物质穿过细胞膜。OxlT 存在于草酸盐降解细菌产草酸杆菌中,有助于控制人体内的草酸盐水平。
过量的草酸会导致肾结石,这是一个痛苦且普遍的健康问题。了解 OxlT 的功能至关重要,但到目前为止,科学家们对其各种结构状态缺乏全面的了解,特别是向内开放构象,这是其运输机制的关键部分。
日本国立自然科学研究院和冈山大学的研究人员利用先进的计算技术来模拟 OxlT 蛋白的动力学。他们利用高斯加速分子动力学(GaMD)和尖端机器学习工具 AlphaFold2 来探索 OxlT 的结构和功能。
图示:OxlT 结构。(来源:论文)
研究人员通过 GaMD 模拟成功预测了 OxlT 的向内开放构象,填补了 OxlT 运输循环中缺失的部分。然后对获得的向内开放的构象进行草酸盐/甲酸盐结合模拟。模拟显示甲酸盐选择性结合到向内开放的构象,而草酸盐在结合位点的入口处被阻断。
该结果为 OxlT 的草酸盐/甲酸盐反向转运功能提供了物理基础:草酸盐被转运到膜的细胞质侧后,甲酸盐(而不是草酸盐)与向内开放的 OxlT 结合,并被转运到膜的周质侧。研究人员将基于物理的 MD 结果与基于机器学习的 AF2 结构预测相结合。野生型序列的 AF2 预测均导致 OxlT 的向外开放构象。
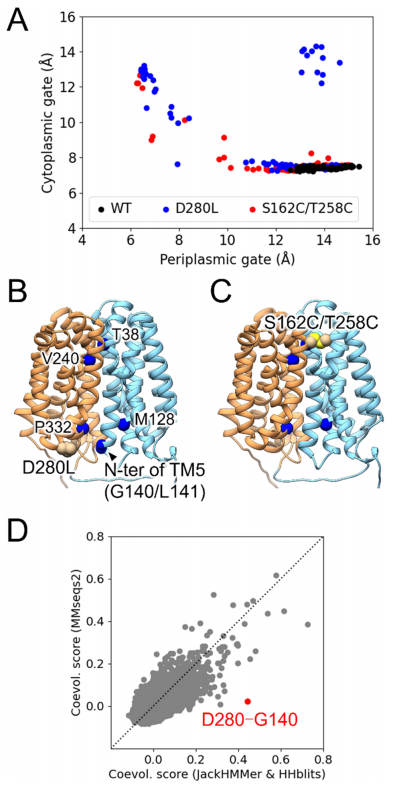
研究人员向参与向内开放状态的关键相互作用的残基引入了单/双突变。在这些突变中,D280L 突变旨在破坏细胞质侧闭塞构象中形成的接触,而 S162C/T258C 突变旨在在向内开放构象的周质侧形成二硫键。他们发现突变使 OxlT 转变为向内开放状态。
尽管之前的研究表明 AF2 无法预测导致天然结构错误折叠或不稳定的点突变,但蛋白质构象变化的情况有所不同,因为 AF2 可以学习 PDB 中同源蛋白质的替代构象。该团队之前的一项研究表明,两个或多个突变可能允许 AF2 探索转运蛋白的替代构象。结合加速 MD 模拟,该团队表示,即使 AF2 中的单个突变也可以改变预测的构象。
此外,研究人员表示 AF2 预测受到输入 MSA 的影响,并且从 MSA 得出的残基协同进化评分可以确定状态转移突变。
图示:AF2 结构预测。(来源:论文)
该团队成功预测了 OxlT 难以捉摸的向内开放构象,这是理解其完整功能循环的重要一步。这种构象表明,在这种状态下,OxlT 更喜欢与甲酸而不是草酸盐结合,这是其在草酸盐管理中发挥作用的一个重要方面。
此外,该研究还确定了对于这种构象转变至关重要的特定氨基酸残基,这一发现可能对理解蛋白质动力学产生更广泛的影响。
这项研究的意义不仅仅局限于单一蛋白质。从这项研究中获得的 *** 和见解,为探索其他蛋白质的动力学提供了一个模板,特别是转运蛋白,它们通常是治疗药物的靶标。
详细了解这些蛋白质可以为各种病症开发更有效的治疗 *** 。此外,这项研究体现了计算生物学与机器学习相结合的力量,这是一个快速发展的领域,有望解开许多生物学中更具挑战性的谜团。
通过填补科学家对 OxlT 蛋白理解的一个关键空白,这项研究不仅有助于肾结石预防方面的潜在进展,而且为未来生物医学研究的突破铺平了道路。
论文链接:https://pubs.acs.org/doi/10.1021/acs.jpclett.3c03052
相关报道:https://phys.org/news/2024-01-molecular-simulation-ai-tool-reveals.html
